Braindecode——开源深度学习电生理数据处理Python包
一. Braindecode 简介
Braindecode 是一个开源的 Python 工具箱,用于解码原始的电生理大脑数据与深度学习模型。它包括数据集获取器、数据预处理和可视化工具,以及几种深度学习的实现 用于分析 EEG、ECoG 和 MEG 的架构和数据增强。
适用于希望使用深度学习和想要处理神经生理学数据的深度学习研究人员。
二. 安装
pip install braindecode
三. 深度学习模型
Braindecode 代码库位于 braindecode/braindecode。
Braindecode 提供了多种深度学习模型,包括:
models_mandatory_parameters = [
("ATCNet", ["n_chans", "n_outputs", "n_times"], None),
("BDTCN", ["n_chans", "n_outputs"], None),
("Deep4Net", ["n_chans", "n_outputs", "n_times"], None),
("DeepSleepNet", ["n_outputs"], None),
("EEGConformer", ["n_chans", "n_outputs", "n_times"], None),
("EEGInceptionERP", ["n_chans", "n_outputs", "n_times", "sfreq"], None),
("EEGInceptionMI", ["n_chans", "n_outputs", "n_times", "sfreq"], None),
("EEGITNet", ["n_chans", "n_outputs", "n_times"], None),
("EEGNetv1", ["n_chans", "n_outputs", "n_times"], None),
("EEGNetv4", ["n_chans", "n_outputs", "n_times"], None),
("EEGResNet", ["n_chans", "n_outputs", "n_times"], None),
("ShallowFBCSPNet", ["n_chans", "n_outputs", "n_times"], None),
(
"SleepStagerBlanco2020",
["n_chans", "n_outputs", "n_times"],
# n_chans dividable by n_groups=2:
dict(chs_info=[dict(ch_name=f"C{i}", kind="eeg") for i in range(1, 5)]),
),
("SleepStagerChambon2018", ["n_chans", "n_outputs", "n_times", "sfreq"], None),
(
"SleepStagerEldele2021",
["n_outputs", "n_times", "sfreq"],
dict(sfreq=100, n_times=3000, chs_info=[dict(ch_name="C1", kind="eeg")]),
), # 1 channel
("TIDNet", ["n_chans", "n_outputs", "n_times"], None),
("USleep", ["n_chans", "n_outputs", "n_times", "sfreq"], dict(sfreq=128)),
("BIOT", ["n_chans", "n_outputs", "sfreq"], None),
("AttentionBaseNet", ["n_chans", "n_outputs", "n_times"], None),
("Labram", ["n_chans", "n_outputs", "n_times"], None),
("EEGSimpleConv", ["n_chans", "n_outputs", "sfreq"], None),
("SPARCNet", ["n_chans", "n_outputs", "n_times"], None),
("ContraWR", ["n_chans", "n_outputs", "sfreq"], dict(sfreq=200)),
("EEGNeX", ["n_chans", "n_outputs", "n_times"], None),
("TSceptionV1", ["n_chans", "n_outputs", "n_times", "sfreq"], dict(sfreq=200)),
("EEGTCNet", ["n_chans", "n_outputs", "n_times"], None),
("SyncNet", ["n_chans", "n_outputs", "n_times"], None),
("MSVTNet", ["n_chans", "n_outputs", "n_times"], None),
("EEGMiner", ["n_chans", "n_outputs", "n_times", "sfreq"], dict(sfreq=200)),
("CTNet", ["n_chans", "n_outputs", "n_times"], None),
("SincShallowNet", ["n_chans", "n_outputs", "n_times", "sfreq"], dict(sfreq=250)),
("SCCNet", ["n_chans", "n_outputs", "n_times", "sfreq"], dict(sfreq=200)),
]
这些模型可以通过 braindecode.models 模块进行访问。
四. 官方文档
Braindecode 官方文档 分为四类:
1. 基础模型构建和训练
- 简单训练MNE epoch
- 简单训练在MNE epoch上进行。
- BCIC IV 2a数据集的裁剪解码
- 在BCIC IV 2a数据集上进行裁剪解码。
- EEG数据的基本脑解码
- 对EEG数据进行基本的脑解码。
- 如何训练、测试和调整你的模型?
- 介绍如何训练、测试和调整模型。
- 使用scikit-learn进行超参数调整
- 使用scikit-learn库进行模型的超参数调整。
- 在假数据上的卷积神经网络回归模型
- 在假数据上构建卷积神经网络回归模型。
- 在PyTorch中训练Braindecode模型
- 介绍如何在PyTorch框架中训练Braindecode模型。
2. 数据加载和组织
- 急切加载与懒加载的基准测试
- 对比急切加载和懒加载的性能。
- 并行化和序列化预处理的基准测试
- 对比并行化和序列化预处理的性能。
- 自定义数据集示例
- 如何使用自定义数据集。
- 加载和保存数据集示例
- 如何加载和保存数据集。
- MNE数据集示例
- 如何使用MNE数据集。
- MOABB数据集示例
- 如何使用MOABB数据集。
- 分割数据集示例
- 如何分割数据集。
- TUH EEG语料库中的多个离散目标
- 如何处理TUH EEG语料库中的多个离散目标。
3. 高级神经网络训练策略
- BCIC IV 4 ECoG数据集上的手指屈曲裁剪解码
- 在BCIC IV 4 ECoG数据集上进行手指屈曲的裁剪解码。
- BCIC IV 2a数据集上的数据增强
- 在BCIC IV 2a数据集上进行数据增强。
- 寻找BCIC IV 2a数据集上的最佳数据增强
- 如何寻找最佳的数据增强方法。
- EEG的相对位置自监督学习
- 如何在EEG数据上进行相对位置的自监督学习。
4. 真实世界数据集的应用示例
- BCIC IV 4 ECoG数据集上的手指屈曲解码
- 在BCIC IV 4 ECoG数据集上进行手指屈曲解码。
- 使用Chambon2018网络的Sleep Physionet数据集睡眠分期
- 如何使用Chambon2018网络对Sleep Physionet数据集进行睡眠分期。
- 使用Eldele2021的Sleep Physionet数据集睡眠分期
- 如何使用Eldele2021对Sleep Physionet数据集进行睡眠分期。
- 使用U-Sleep网络的Sleep Physionet数据集睡眠分期
- 如何使用U-Sleep网络对Sleep Physionet数据集进行睡眠分期。
- 处理大型EEG数据资源(TUH EEG语料库)
- 如何处理TUH EEG语料库中的大规模EEG数据。
这些示例提供了Braindecode库在不同场景下的应用,从基础的模型构建到高级的神经网络训练策略,以及如何在真实世界的数据集上应用这些技术。
五. 其他资源——SSVEP解码深度学习算法仓库 DL_Classifier
1. DL_Classifier简介
稳态视觉诱发电位(SSVEP)是指当受试者持续注视以固定频率闪烁或翻转的刺激时,在大脑的枕叶-额叶区域中诱发的与刺激频率相关的电生理信号。与P300、运动意向(MI)等EEG信号相比,SSVEP具有更高的信噪比,可以产生更高的ITR,使其成为长期以来最具潜力的EEG范式之一。然而,传统的信号处理算法在解码SSVEP信号时依赖于手动特征提取,在各种极端情况下(数据长度短、刺激数量大、校准数据量小)表现不佳,无法满足行业的实际需求。深度学习技术是人工智能的主要分支之一,已经在计算机视觉、自然语言处理、推荐系统等各个领域的研究中得到应用。由于其强大的特征表达能力和极高的灵活性,它颠覆了这些领域的算法设计思想,并取得了显著的成果。
近年来,深度学习技术逐渐受到BCI领域研究人员的青睐。DL_Classifier提供了用于复现SSVEP信号的基于深度学习的识别方法。复现的方法包括EEGNet [1]-[2]、C-CNN [3]、FBtCNN [4]、ConvCA [5]、SSVEPNet [6]、SSVEPformer [7]和DDGCNN [8],并使用了一个12类公共数据集 [9] 进行评估。
DL_Classifier 的仓库为:
https://github.com/YuDongPan/DL_Classifier/
https://github.com/LSYhhhh/DL_Classifier
2. 模型介绍
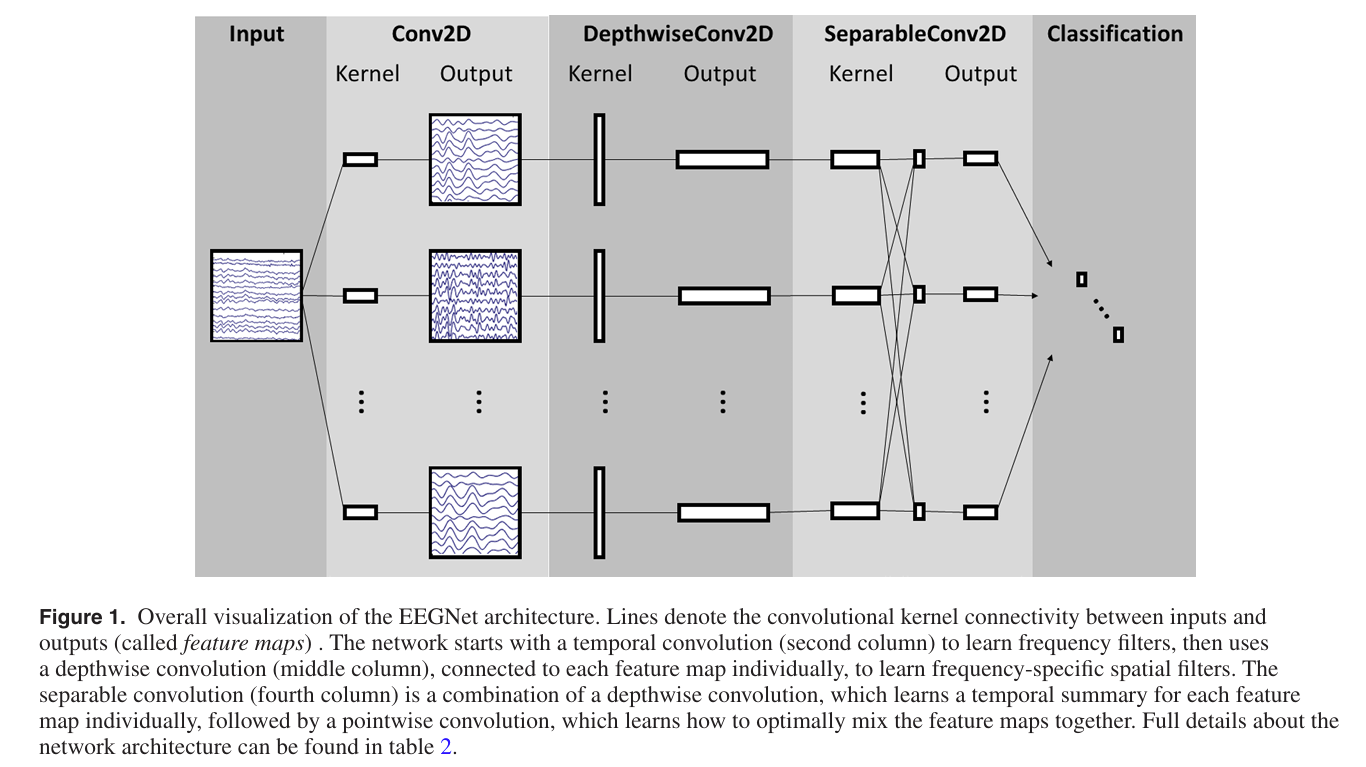
EEGNet: EEGNet是一种专门设计用于处理EEG信号数据的卷积神经网络模型,它接收时间域EEG数据作为网络输入。EEGNet由4层组成。第一层是卷积层,用于模拟对每个通道进行带通滤波。第二层是空间滤波层,用于对每个通道的数据进行加权,通过深度卷积实现。第三层是用于提取类别信息的单独卷积层。第四层是用于分类的全连接层。自提出以来,EEGNet已被用于各种EEG任务,如运动意向、P300、SSVEP等。
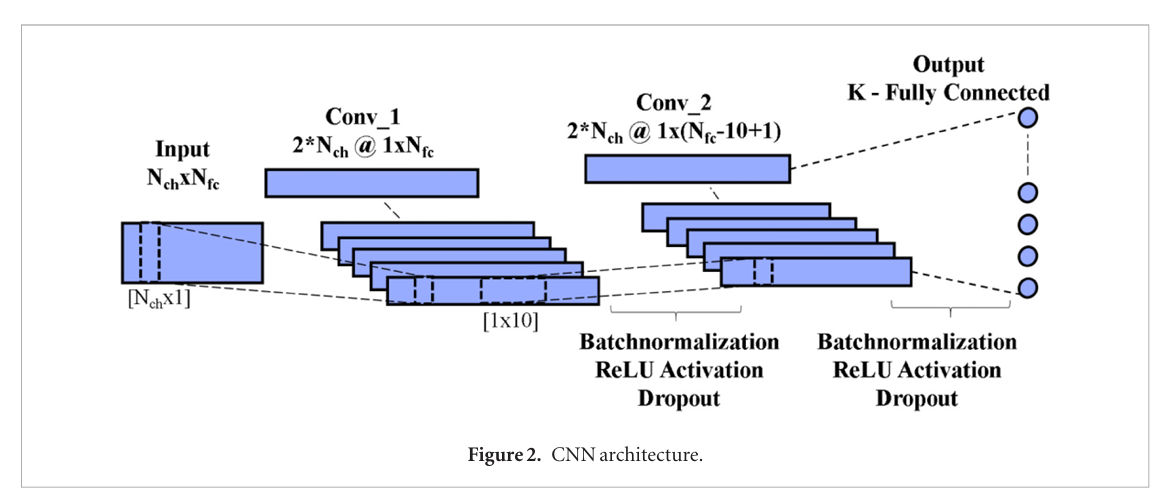
CCNN: CCNN也是一种卷积神经网络模型。不同的是,C-CNN使用富含幅度和相位信息的频域数据作为网络输入。值得注意的是,为了获得网络的输入,CCNN使用基于填充的FFT算法,可以在任何数据长度下获得220个数据点,包括110个实部数据和110个虚部数据。CCNN网络结构由两个卷积层和一个全连接层组成。第一个卷积层是空间滤波层,第二个卷积层是时间滤波层,全连接层用于分类。CCNN的出现表明,频谱数据有助于SSVEP分类。
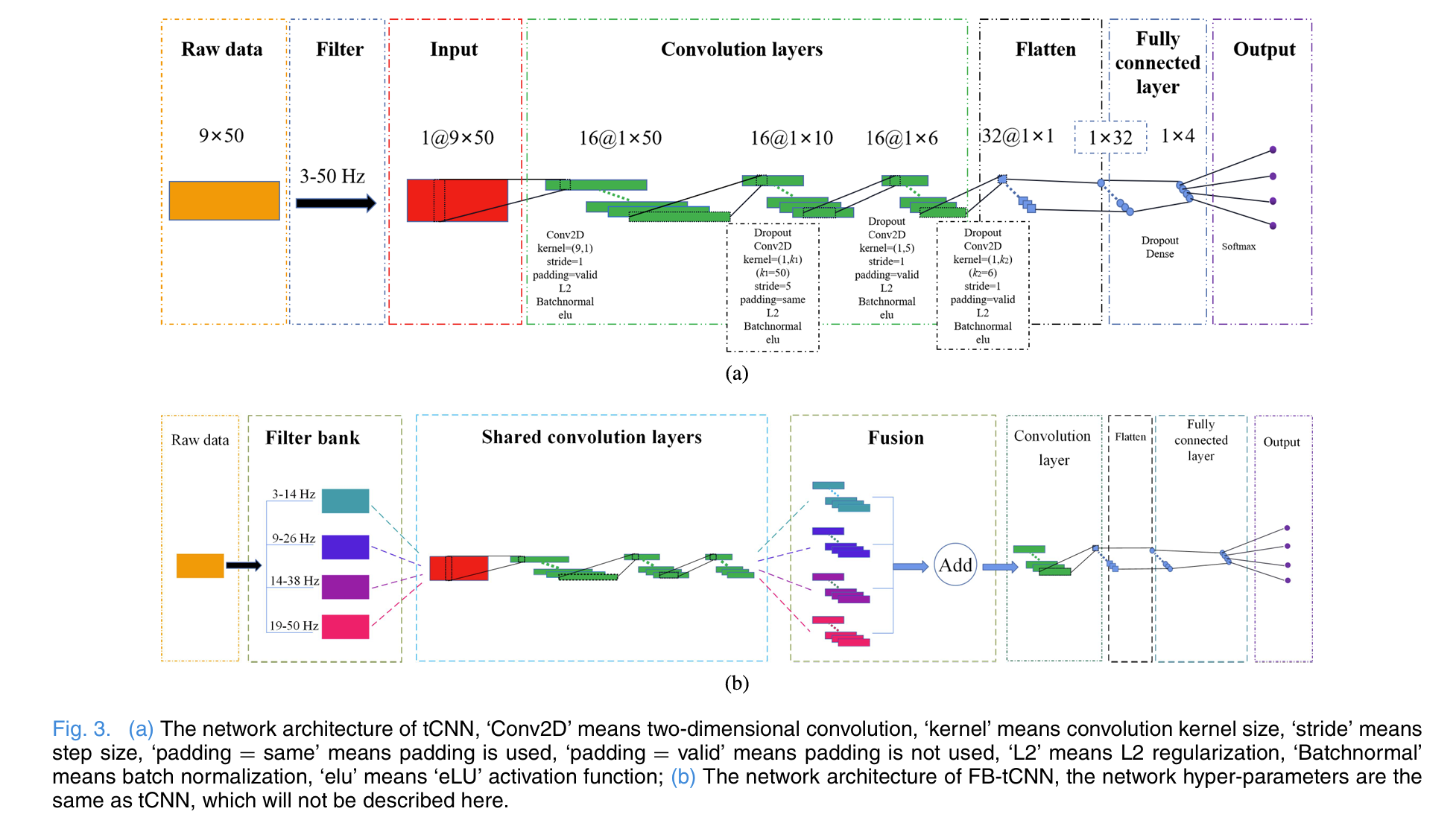
FBtCNN: 在频域输入范式下,短时窗口下的特征可能不明显,每个通道的时间差异可能被忽略。考虑到谐波信息中嵌入了大量有关频率识别的有效信息,研究人员提出了一种基于滤波器组技术的CNN架构,称为FBtCNN。FBtCNN使用时间域信号作为网络输入,并使用滤波器组来融合来自不同频段的特征信息,以提高网络的分类性能。
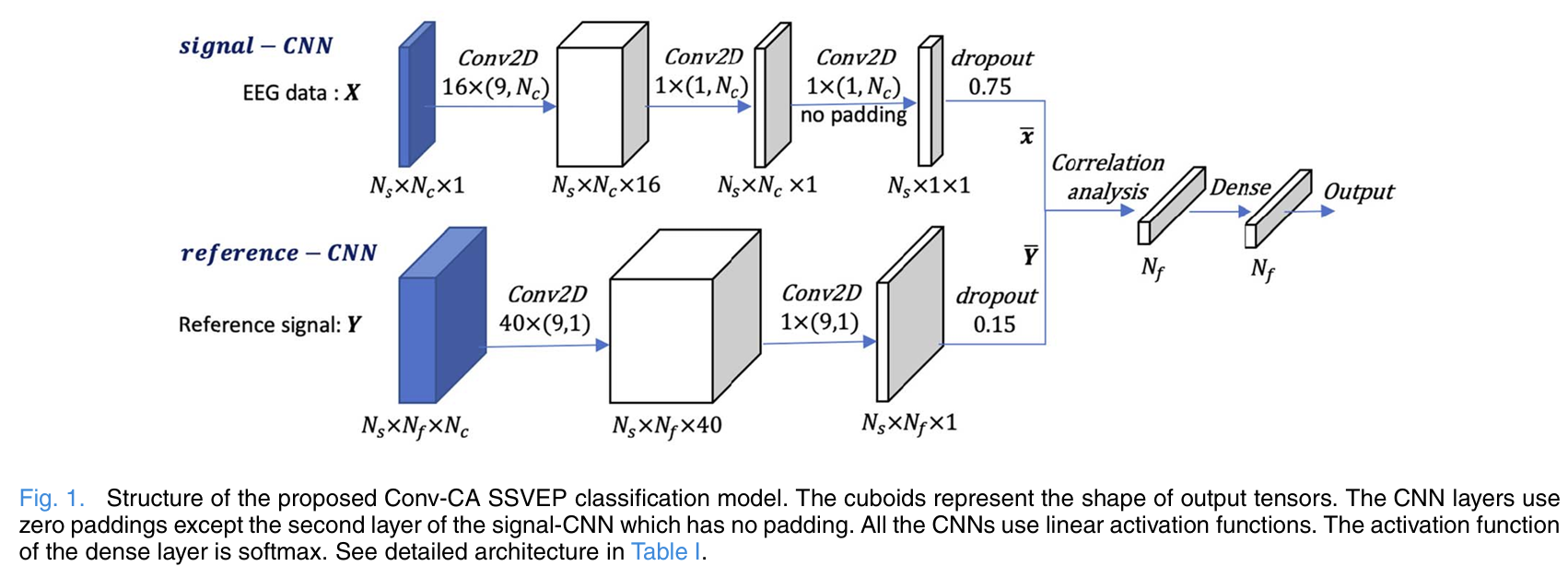
ConvCA: 来自不同电极通道的SSVEP信号之间存在非线性关系,多通道EEG信号的线性组合可能不是分类SSVEP的最准确解决方案。为了进一步提高SSVEP-BCI的性能,研究人员提出了一种基于CNN的非线性相关性分析网络,称为ConvCA。ConvCA有两个分支,一个是信号网络,将多通道EEG信号降级为单通道EEG信号,另一个是参考网络,将多通道参考信号解释为单通道参考信号。ConvCA通过分析信号网络输出和参考网络输出之间的相关性来完成SSVEP的分类过程。
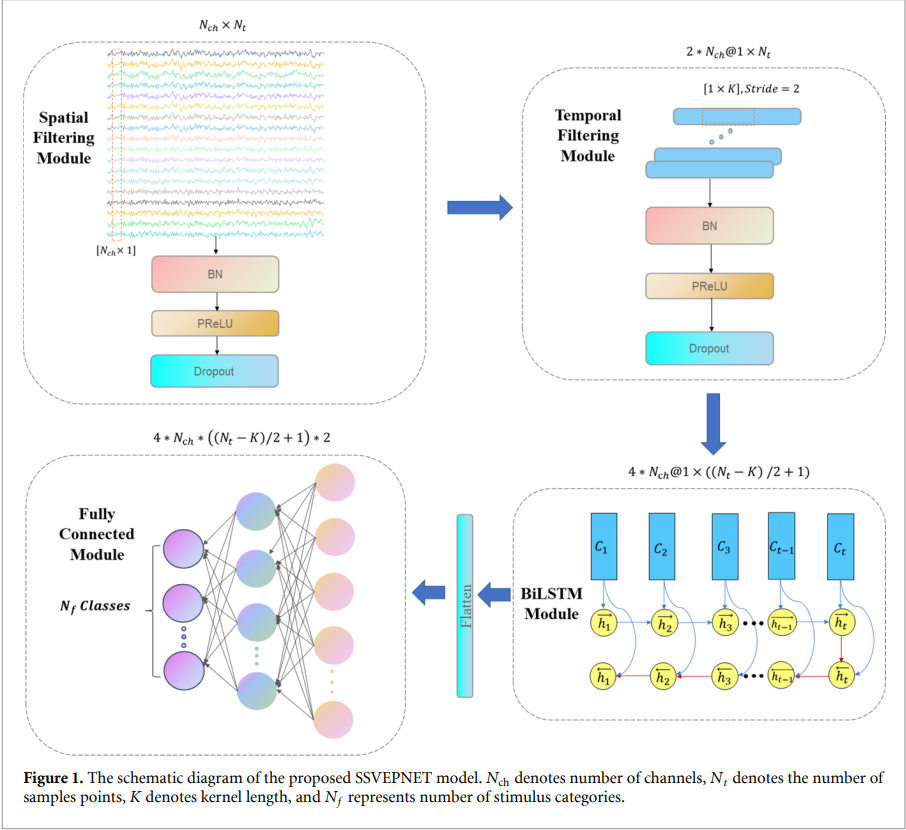
SSVEPNet: SSVEPNet是卷积神经网络和长短期记忆网络的混合网络模型,接受时间域数据作为网络输入。SSVEPNet由三部分组成:卷积神经网络、长短期记忆网络和全连接网络。其中,CNN用于提取EEG的时空特征,双向LSTM用于基于时空特征之间的依赖关系进行编码,由三个全连接层组成的全连接网络用于最终分类。此外,为了在网络训练过程中抑制可能的过拟合,网络实现过程中结合了谱归一化和基于标签的平滑技术,进一步提高了模型的泛化能力。SSVEPNet即使只有少量校准数据,仍然可以实现非常好的分类性能,并且这一特性已在4类、12类和40类数据集上得到验证。
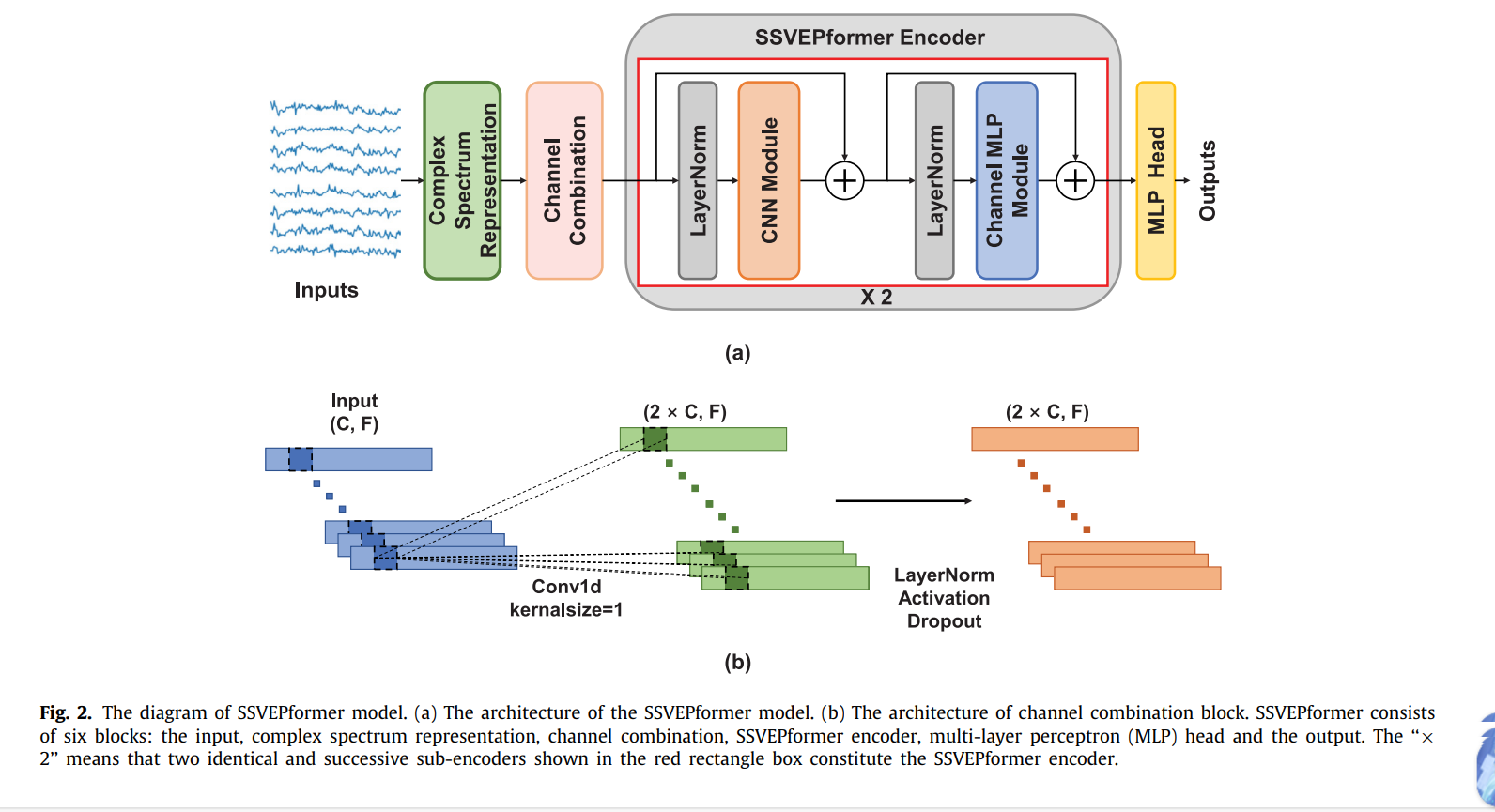
SSVEPformer: 基于注意力机制的Transformer已经应用于CV和NLP等多个领域,并取得了引起全球关注的显著成果。近年来,Transformer逐渐被引入到EEG信号的解码和分析中,揭示了Transformer在SSVEP-BCI系统中的潜在应用。由于当前SSVEP解码算法对大量受试者校准数据的依赖,研究人员提出了基于Transformer的SSVEP分类网络SSVEPformer,以实现零校准高性能网络。SSVEPformer和C-CNN的网络输入保持一致,使用频域信号作为输入,并结合CNN和MLP网络完成Transformer的特征编码和注意力学习过程。SSVEPformer可以在跨受试者实验中实现显著的分类性能,明显优于其他分类网络。此外,结合滤波器组技术,研究人员提出了网络的改进版本FB-SSVEPformer,以进一步增强网络性能。
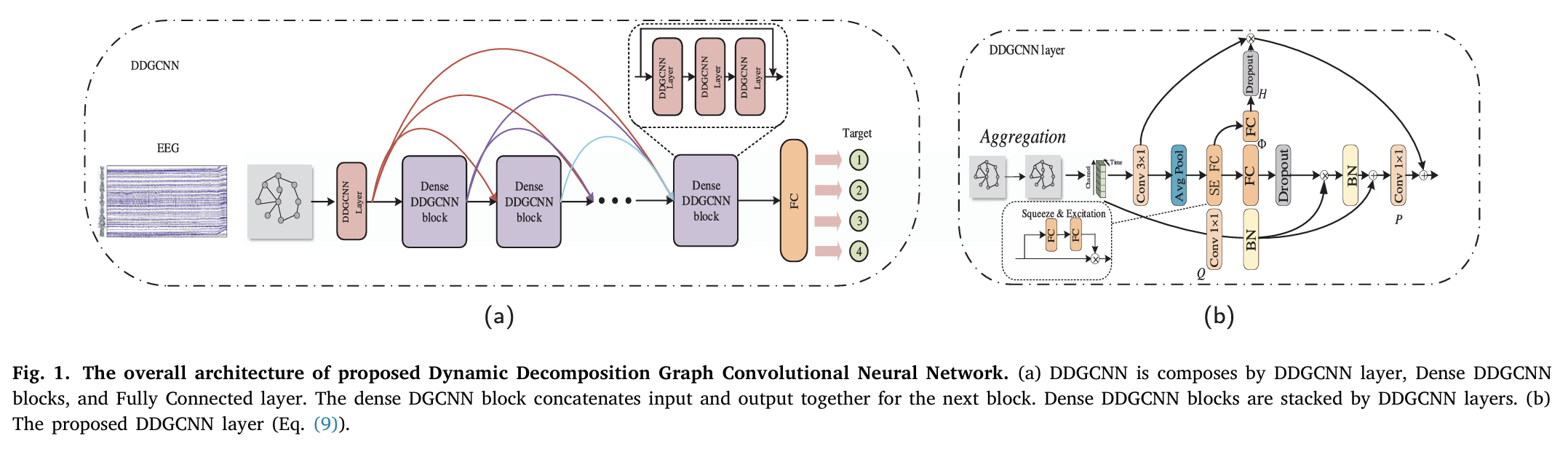
DDGCNN: 在多通道EEG分类过程中,多通道脑电图(EEG)数据的处理由于其非欧几里得特性而引入了挑战,需要考虑通道间的拓扑关系,因此需要考虑通道间的拓扑关系。研究人员引入了用于SSVEP EEG信号分类的动态分解图卷积神经网络(DDGCNN)。DDGCNN结合了逐层动态图,以解决图卷积网络(GCN)中的过度平滑问题,采用密集连接机制来减轻梯度消失问题。此外,DDGCNN通过图动态融合增强了GCN中固有的传统线性变换,从而提高了特征提取和自适应聚合能力。实验结果表明,DDGCNN在学习和提取EEG拓扑结构特征方面的有效性。
3. 代码运行环境
- 设置虚拟环境,使用python 3.8或更新版本
- 安装依赖
pip install -r Resource/requirements.txt
- 编辑配置文件(etc/config.yaml),选择要运行的算法。
- 编辑配置文件,设置训练过程的参数和算法的特定超参数。
- 运行
python Classifier_Test.py
4. 参考文献
[1] Lawhern V J, Solon A J, Waytowich N R, et al. EEGNet: a compact convolutional neural network for EEG-based brain–computer interfaces[J]. Journal of neural engineering, 2018, 15(5): 056013. https://iopscience.iop.org/article/10.1088/1741-2552/aace8c/meta
[2] Waytowich N, Lawhern V J, Garcia J O, et al. Compact convolutional neural networks for classification of asynchronous steady-state visual evoked potentials[J]. Journal of neural engineering, 2018, 15(6): 066031. https://iopscience.iop.org/article/10.1088/1741-2552/aae5d8/meta
[3] Ravi A, Beni N H, Manuel J, et al. Comparing user-dependent and user-independent training of CNN for SSVEP BCI[J]. Journal of neural engineering, 2020, 17(2): 026028. https://iopscience.iop.org/article/10.1088/1741-2552/ab6a67/meta
[4] Ding W, Shan J, Fang B, et al. Filter bank convolutional neural network for short time-window steady-state visual evoked potential classification[J]. IEEE Transactions on Neural Systems and Rehabilitation Engineering, 2021, 29: 2615-2624. https://ieeexplore.ieee.org/abstract/document/9632600/
[5] Li Y, Xiang J, Kesavadas T. Convolutional correlation analysis for enhancing the performance of SSVEP-based brain-computer interface[J]. IEEE Transactions on Neural Systems and Rehabilitation Engineering, 2020, 28(12): 2681-2690. https://ieeexplore.ieee.org/abstract/document/9261605/
[6] Pan Y, Chen J, Zhang Y, et al. An efficient CNN-LSTM network with spectral normalization and label smoothing technologies for SSVEP frequency recognition[J]. Journal of Neural Engineering, 2022, 19(5): 056014. https://iopscience.iop.org/article/10.1088/1741-2552/ac8dc5/meta
[7] Chen J, Zhang Y, Pan Y, et al. A Transformer-based deep neural network model for SSVEP classification[J]. Neural Networks, 2023, 164: 521-534. https://www.sciencedirect.com/science/article/abs/pii/S0893608023002319
[8] Zhang S, An D, Liu J, et al. Dynamic decomposition graph convolutional neural network for SSVEP-based brain–computer interface[J]. Neural Networks, 2024, 172: 106075.https://www.sciencedirect.com/science/article/abs/pii/S0893608023007360
[9] Nakanishi M, Wang Y, Wang Y T, et al. A comparison study of canonical correlation analysis based methods for detecting steady-state visual evoked potentials[J]. PloS one, 2015, 10(10): e0140703. https://journals.plos.org/plosone/article?id=10.1371/journal.pone.0140703

